ニュース・イベント
News & Events
ニュース・イベント
News & Events
ニュース
News
2016年6月10日
ES細胞と遺伝子ネットワークを用いた、高精度の化合物毒性予測システムの構築
ポイント
- ES細胞を用いることで、数か月~数年後に現れる化合物の毒性を予測できる。
- 数理的な遺伝子ネットワーク注1構築と機械学習を応用することで、化合物の構造により毒性を予測するという従来法と比べ、高い確率で毒性を予測できるシステムを構築した。
山根順子研究員(京都大学CiRA)と藤渕航教授(京都大学CiRA増殖分化機構研究部門)は、東京大学大学院医学系研究科疾患生命工学センター、産業技術総合研究所生命情報工学研究センター、国立環境研究所環境リスク・健康研究センター及び群馬大学情報工学科との共同研究で、ヒトES細胞、遺伝子ネットワークと機械学習技術を活用した高精度の、化合物の毒性予測システムを構築しました。
この研究成果は2016年5月20日(英国時間)に英国科学誌「Nucleic Acids Research」でオンライン公開されました。
創薬において、化合物の毒性評価は必須ですが、毒性評価を人体で直接研究することは許されておらず、従来、動物モデルでの試験を行ってきました。しかしながら、動物の体内の作用と人体での作用は異なる場合も少なくなく、動物実験を経て承認された薬物が人でがんや精神疾患など重篤な作用をもたらすことがあります。そのため、ヒトの細胞や組織を用いた毒性評価系の構築が望まれていました。近年では、iPS細胞のようなヒトの細胞が入手できるようになり、それを特定の細胞種に分化させてから化合物を添加し、その細胞の挙動を見ることで毒性評価を行っていますが、ばらつきのない標準的な、分化した細胞を作る必要があります。そこで、藤渕教授らのグループはES細胞に化合物を添加し、それによって生じる遺伝子発現量の変化をもとに遺伝子ネットワークを構築、機械学習の技術を組み合わせることで、精度の高い毒性予測のしくみを構築しました。
1) 遺伝子ネットワークを利用した化合物毒性予測システムを構築した
今回の研究では、神経毒性(9つ)・遺伝的発がん性(5つ)・非遺伝的発がん性(6つ)・不明(2つ)にカテゴリー分けされた22の化合物を対象に、毒性予測システムの構築を試みました。その中の10化合物を試験的に用いて、どの遺伝子の働きを調べれば、毒性を予測できるのかを調べました。まずES細胞に化合物を添加し、遺伝子の発現量をマイクロアレイ注2で測定しました。そして、添加前と比較して発現量の差が大きかった、転写因子注3をコードする遺伝子を候補として絞り込みました。さらに、検出できないほどES細胞でほとんど発現していない遺伝子を除外した結果、10の遺伝子に絞り込みました。この10遺伝子が、化合物毒性を予測する上で重要な代表遺伝子となります。
次に、対象の22の化合物をES細胞にそれぞれ添加し、10の代表遺伝子の発現量を計測しました。そして、そのデータをもとに数理モデルを用いて、遺伝子ネットワークを推定しました。これにより、ある化合物を添加すると細胞内の遺伝子の発現の上昇あるいは低下が、他の遺伝子の発現量にどのように影響を及ぼすかを示すことができます。
遺伝子ネットワークのパターンを化合物間で比較したところ、それぞれで違いが見られたものの、カテゴリー別に化合物の遺伝子ネットワークパターンを重ね合わせると、同カテゴリーでは共通の遺伝子間の作用経路があるなど、類型化できることが分かりました。さらに、カテゴリー間を比較したところ、遺伝子ネットワークの共通パターンの相違、つまり反応の違いがあることが明らかになりました。
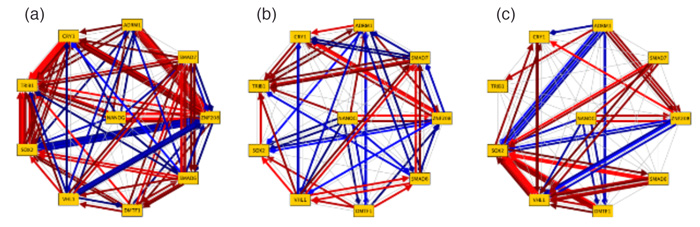
図. カテゴリー別に、遺伝子ネットワークパターンを類型化したもの
赤い矢印は、ある遺伝子の発現上昇が、下流の遺伝子発現を上昇させるといった正の制御を、 青い矢印はある遺伝子の発現上昇が、下流の遺伝子発現を抑制するといった負の制御を示している。
(a) 神経毒性(9化合物)、(b) 遺伝的発がん性(5化合物)、(c) 非遺伝的発がん性(6化合物)
この遺伝子ネットワークのパターンを機械学習させ、その違いを利用して、改めて、それぞれの化合物が本来のカテゴリーに属すると予測されるかを調べたところ、ランダムあるいは従来の、化合物の構造を数値化することで毒性を予測するシステムよりも高精度に予測できることが示されました。
2) 化合物の毒性予測を行った
今回選定した22の化合物のうち、機械学習に用いなかった、カテゴリーが未確定の2種の化合物(ビスフェノールAとペルメトリン)について、選定した10遺伝子の遺伝子発現量を測定し、遺伝子ネットワークを構築しました。すると、ビスフェノールAはダイオキシン類化合物に近いパターンで非遺伝的発がん性のカテゴリーに分類され、ペルメトリンは神経毒性と非遺伝的発がん性のカテゴリーに分類されました。先行研究に、ビスフェノールAの胎児期曝露と生後の発がん性の関連性やペルメトリンと神経毒性の関連を示唆するという報告があり、本予測と合致しました。ペルメトリンの発がん性を示す先行研究はまだなく、この点に関してはさらなる研究が必要であると思われます。
本研究では、分化した細胞を用いず、ES細胞のみから精度高く化合物の毒性を予測できるシステムを構築しました。
この毒性予測システムは、従来の、化合物の構造から予測するシステムよりも高い精度で化合物の毒性を予測できることが示されました。今後は、遺伝子ネットワークだけでなく細胞の形態などの指標を増やすことでさらに精度を高めたり、他の組織特異的な毒性(例えば骨への毒性など、他のカテゴリーの毒性)の予測にも対応できるようにすることを目指します。また、将来的には様々な人のiPS細胞を利用し、毒性評価予測を行うことで個別化医薬へ貢献することが期待されます。
- 論文名
"Prediction of Developmental Chemical Toxicity based on Gene Networks of Human Embryonic Stem Cells" - ジャーナル名
Nucleic Acids Research - 著者
Junko Yamane1,2, Sachiyo Aburatani3, Satoshi Imanishi2, Hiromi Akanuma4, Reiko Nagano4, Tsuyoshi Kato5, Hideko Sone4, Seiichiroh Ohsako2 and Wataru Fujibuchi1,3 - 著者の所属機関
- 京都大学 iPS細胞研究所(CiRA)
- 東京大学 大学院医学系研究科 疾患生命工学センター
- 産業技術総合研究所 生命情報工学研究センター
- 国立環境研究所 環境リスク・健康研究センター
- 群馬大学 情報工学科
本研究は、下記機関より資金的支援を受けて実施されました。
- 厚生労働省 科学研究費補助金(代表:大迫、分担:藤渕、曽根)
- 再生医療実現拠点ネットワークプログラム iPS細胞研究中核拠点
- 文部科学省/日本学術振興会 科学研究費助成事業(15H01749、代表:曽根、分担:藤渕)
- 環境省 環境研究総合推進費(代表:曽根、分担:大迫)
<用語説明>
-
注1)遺伝子ネットワーク
遺伝子どうしの制御のネットワーク。マイクロアレイで得た遺伝子発現のデータから数理モデルを用いて、推定・構築できる。
-
注2)マイクロアレイ
一度に膨大な数のDNAやRNA、タンパク質を網羅的に検査することができる解析技術。
- 注3)転写因子
タンパク質合成は、DNA上の遺伝子を鋳型としてメッセンジャーRNAが転写され、このメッセンジャーRNAが核外のリボソーム上で翻訳される過程で成り立っている。転写因子は、転写開始に関わるタンパク質因子で、DNAに結合して働くものや因子間の相互作用によって機能するものがある。