研究活動
Research Activities
研究活動
Research Activities
研究成果
Publications
2022年4月11日
遺伝暗号開始コドンの修飾がmRNAの性能を変える 〜修飾塩基によりmRNAからタンパク質の作られやすさが変わる新しいメカニズムを発見!〜
ポイント
- 細胞でのタンパク質合成(翻訳)は、通常、mRNA上の塩基配列「AUG」から始まるが、稀に他の塩基配列から始まることもある。この始まりやすさ(翻訳開始効率)が、塩基の化学修飾によって変化することを明らかにした。
- さらに、分子シミュレーションにより、RNA分子の結合しやすさが化学修飾により変化すること、それが翻訳開始効率の変化をよく説明できることを示した。
- 化学修飾されたmRNAは、最近、新型コロナウイルスワクチンにも応用され脚光を浴びた。本来は使われないAUG以外の開始コドンと化学修飾によるリコーディングを組み合わせることで、mRNA医薬をより安全にできる可能性がある。
mRNAはタンパク質の「設計図」のコピーとして働く分子です。その情報を記録する塩基には、ときに化学的な「修飾」が加わることがあります。
広島大学大学院統合生命科学研究科の浅野桂特任教授(広島大学健康長寿研究拠点メンバー、カンザス州立大学生物学科教授兼任)、京都大学iPS細胞研究所の藤田祥彦研究員と齊藤博英教授、立命館大学生命科学部の亀田健客員研究員と冨樫祐一教授らの共同研究グループは、mRNAの修飾がタンパク質合成の効率を変化させることを明らかにしました。近年、人工的な修飾mRNAが、ワクチンなど医薬にも用いられており、その改良に応用できる可能性があります。
本研究成果は、国際学術誌「Science Advances」に4月8日(金)(米国時間)に掲載されました。
リボソーム(注1)は、細胞内でmRNA(注2)の情報に基づき、翻訳(タンパク質合成)を行います。mRNAは4種類の塩基(A・C・G・U)を用いて情報を記録しており、3個の塩基(コドン)で1つのアミノ酸を指定します。1塩基ずれたところから翻訳を始めると、全く異なるアミノ酸配列になるため、始める場所は厳密に決める必要があります。通常、これは開始コドンと呼ばれる塩基配列「AUG」で指定されます。
しかし、これには例外がありました。バクテリアなどの原核生物では、「GUG」や「UUG」からの翻訳開始もみられます。ヒトなどの真核生物では「AUG」以外からの翻訳開始は稀ですが、その中では「CUG」からの翻訳開始が比較的多く、原核生物とは異なる傾向となっています。このように、翻訳の始まりやすさ(翻訳開始効率)は塩基配列によるものの、それが決まるメカニズムは謎に包まれていました。
一方で、核酸塩基は、酵素などを用いて化学的に変化させることができます。mRNAにもそのようにして少しだけ分子構造を変化(修飾)させた塩基がみられます(図(A)参照)。また、修飾塩基を含んだmRNAを人工的に合成することもできます。例えば、iPS細胞の作製や、最近では新型コロナウイルス(SARS-CoV2)ワクチンにも応用されています。これは主に、外来のmRNAへの免疫を回避する目的で用いられています。しかし、修飾によって翻訳開始効率も変化するのであれば、応用における効率化や制御の新たな手段にもなり得ます。
本研究では、mRNAの開始コドン部分を改変したときのタンパク質合成量の増減を体系的に解析し、塩基の修飾が翻訳開始効率を変化させることを明らかにしました。
浅野桂特任教授ら、広島大学大学院、カンザス州立大学生物学科のチームは、蛍光タンパク質の遺伝子を用いて、これらの箇所を様々に改変したmRNAを精製しました。このmRNAを細胞に導入すると、蛍光の強さから、つくられたタンパク質の相対量を知ることができます。修飾塩基の導入でmRNAの分解の速さが変わらないことが示唆されたため、タンパク質の量の変化から翻訳開始効率の変化が分かることになります。
計測の結果、「GUG」や「CUG」の「U」を、「ψ」(シュードウリジン、図(A))や「1mψ」(N1-メチルシュードウリジン、mRNAワクチンにも利用されている)に変えると、翻訳開始効率が上昇する一方で、「CUG」の「C」を「5mC」(5-メチルシチジン、図(A))や「5hmC」(5-ヒドロキシメチルシチジン)に変えると、翻訳開始効率は低下することが明らかになりました。京都大学iPS細胞研究所(CiRA)の藤田祥彦助教と齊藤博英教授は、ヒト由来の3種類の細胞で計測した結果、この傾向が、iPS細胞やがん細胞といった細胞の種類によらず、共通していることが確認されました。
さらに、この変化が生じる原因を明らかにするため、立命館大学生命科学部の亀田健客員研究員と冨樫祐一教授(理化学研究所兼任)は、分子動力学計算(注3)を用いた解析を行いました。翻訳開始効率が高い塩基配列や修飾ほど、リボソームの中でmRNAとそれを読み取るtRNA(注4)とが結合しやすい傾向がみられました。結合した際の分子構造から、修飾による効率変化の原因も示唆されました。
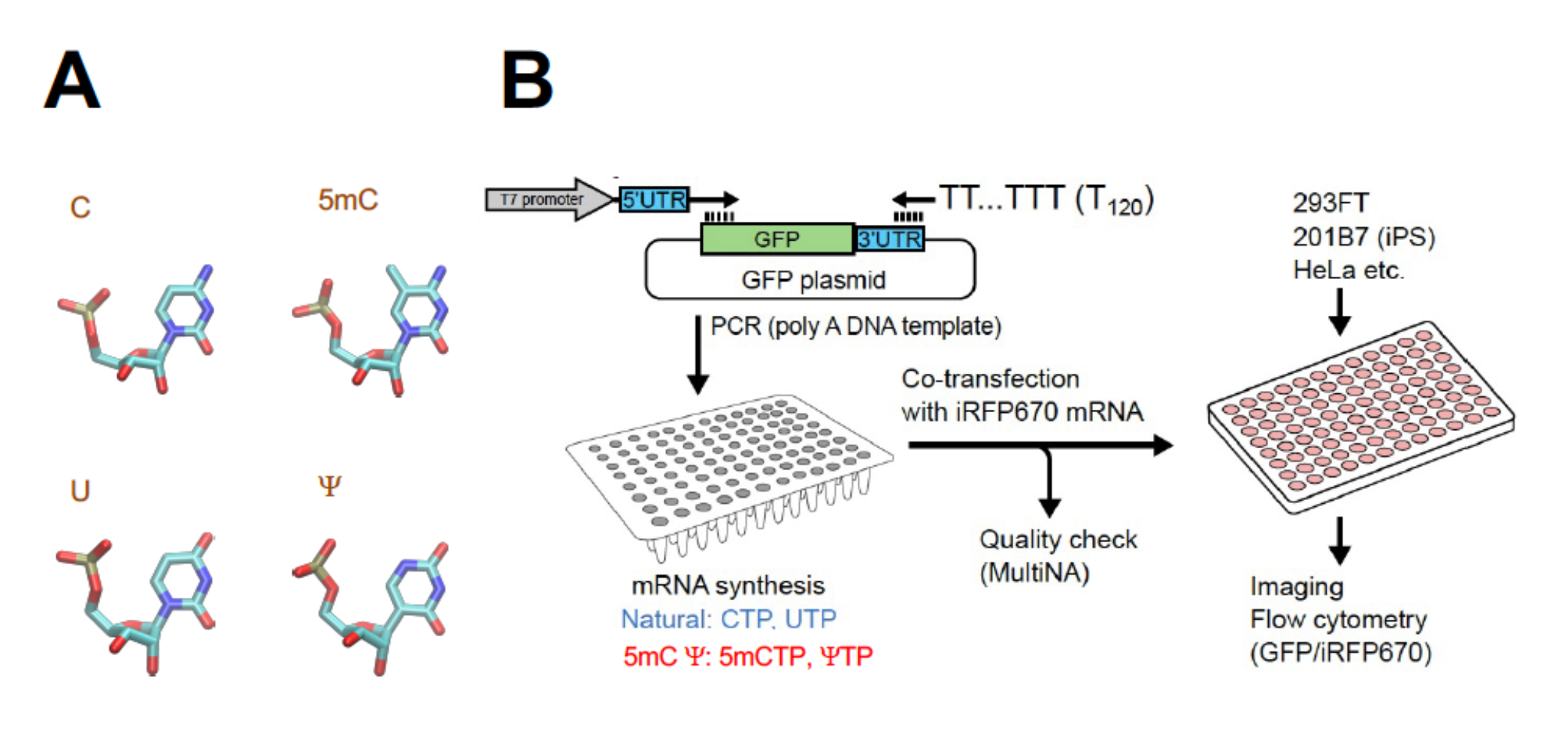
(A)RNAの塩基と修飾の例(水色:炭素、青:窒素、赤:酸素、黄:リン。水素は略)。
例えば5mCではCの5-位の水素に代えてメチル基が導入されている。(B)実験の概要。
本研究で示されたように、修飾mRNAとtRNAとの間の親和性が翻訳開始効率を左右するのであれば、新たな修飾を合理的に設計して翻訳を制御できると期待されます。特にmRNAの医薬応用などにおいて、タンパク質発現を効率的にできる可能性があります。開始コドンをAUGでないタイプに変えて化学修飾することで、逆転写過程によってゲノムに組み込まれたとしても予期せぬ翻訳を引き起こす心配がなくなります。このような開始コドンの化学修飾は、新たなmRNA医薬の開発につながる可能性があります。
- 論文名
Translational recoding by chemical modification of non-AUG start codon ribonucleotide bases - ジャーナル名
Science Advances - 著者
Yoshihiko Fujita+, Takeru Kameda+, Chingakham Ranjit Singh+, Whitney Pepper, Ariana Cecil,
Madelyn Hilgers, Mackenzie Thornton, Izumi Asano, Carter Moravek, Yuichi Togashi*, Hirohide Saito*, Katsura Asano*
+Equal contribution, *Corresponding authors
本研究の遂行にあたり、日本学術振興会(科研費JP18K19963, JP18KK0388, JP20H05626、外国人研究者招へい事業)、平和中島財団、京都大学iPS細胞研究所(iPS細胞研究基金)、カンザス州立大学(Johnson Cancer Research Center Innovative Award, Travel Award; Faculty Development Award)、アメリカ国立衛生研究所(Grant GM124671)、アメリカ国立科学財団(Research Grant 1412250)の助成を受けました。シミュレーションには九州大学情報基盤研究開発センター研究用計算機システムITO、理化学研究所共同利用計算機HOKUSAIを使用しました。
(注1)リボソーム
細胞内でタンパク質を合成する役割を持つ分子複合体。RNA(リボ核酸)とタンパク質からなる巨大な複合体である。
(注2)mRNA(メッセンジャーRNA、伝令RNA)
遺伝子の一時的なコピーとして、リボソームで合成されるタンパク質のアミノ酸配列を指定する役割を持つRNA。
(注3)分子動力学計算
分子の中の各原子に働く力を近似的に求め、運動方程式に基づいて各原子の動きを計算することで、分子の構造変化や動きをシミュレーションする方法。分子の構造変化を、実験では直接観察できない細部まで推定することができる。非常に多くの計算を必要とするのが弱点であるが、本研究では、適応バイアス力法と呼ばれる手法で効率化するなどの工夫により適用を可能にした。
(注4)tRNA(トランスファーRNA、転移RNA)
mRNAで指定されたアミノ酸を配置するアダプターの役割を持つRNA。酵素により特定のアミノ酸と結合される部位と、mRNAのコドンと対をなす部位(アンチコドン)とを持っており、指定のアミノ酸をリボソームに供給する。本研究では、このコドン-アンチコドン間の塩基対形成に注目し、塩基間の距離を指標としてシミュレーションにより結合親和性を評価した。