研究活動
Research Activities
研究活動
Research Activities
研究成果
Publications
2023年5月19日
オミクロンXBB株の進化経路とウイルス学的特性の解明
―遺伝子組換えによるさらなる免疫逃避能力の獲得―
ポイント
- XBB株は、2種類のオミクロン株(オミクロンBJ.1株とBM.1.1.1株)がスパイクタンパク質の受容体結合部位(RBD)で遺伝子組換えすることによって高い実効再生産数(流行拡大能力)を獲得したことを明らかにした。
- オミクロンXBB株は、高い液性免疫からの逃避能、細胞への侵入効率、そして合胞体形成活性を遺伝子組換えにより獲得した。
- ハムスターモデルにおけるオミクロンXBB株の病原性は、オミクロンBA.2.75株と比較してやや弱かった。
出口清香 大学院生(CiRA増殖分化機構研究部門)、高山 和雄 講師(CiRA同部門)、佐藤 佳 教授(東京大学医科学研究所システムウイルス学分野)、研究コンソーシアム「The Genotype to Phenotype Japan (G2P-Japan)」注1は、2022年9月頃からインドを中心に流行を拡大したオミクロン株(B.1.1.529, BA系統)注2XBB株の進化の軌跡とウイルス学的特性を明らかにしました。進化系統解析により、オミクロンXBB株は、2種類のオミクロン株(オミクロンBJ.1株とBM.1.1.1株[BA.2.75株の子孫株])がスパイクタンパク質注3の受容体結合部位(RBD)注4において遺伝子組換え注5を起こしたことで生じた変異株であること、そしてオミクロンXBB株は2022年の夏頃に出現したことを示しました。
さらに本研究では、オミクロンXBB株についての詳細なウイルス学的実験を行いました。オミクロンXBB株は、祖先株であるBA.2.75株およびBA.2.75株の祖先株であるBA.2株よりも高い液性免疫注6に対する逃避能、感染受容体であるACE2注7結合能、そして感染性を遺伝子組換えにより獲得したことが明らかとなりました。一方で、オミクロンXBB株の実験動物モデルにおける病原性はオミクロンBA.2.75株に比べ、やや弱いことが明らかとなりました。
この研究成果は2023年5月16日(英国時間)に「Nature Communications」でオンライン公開されました。
ウイルス感染症の制御が難しい一因は、ウイルスが変異を獲得し進化することにあります。例えば、新型コロナウイルス(SARS-CoV-2)は流行の過程で著しく多様化し、「変異株」と呼ばれる様々な特性を持ったウイルスが出現してきました。現在でも、ワクチン接種や自然感染により獲得された液性免疫に対する高い逃避能を持った変異株が次々と出現してきており、医学・公衆衛生上の問題となっています。SARS-CoV-2の研究により、ウイルスの進化と流行の原理を理解することができれば、新型コロナウイルス感染症(COVID-19)だけでなく、将来のパンデミックを含めた様々な感染症の制御に繋がる知見を得ることができると期待されます。
2021年末に出現したオミクロン株(B.1.1.529、BA系統)は、流行の過程で著しく多様化し、様々なオミクロン亜株(オミクロンBA.2株、BA.5株、BA.2.75株、BQ.1.1株、XBB株など)が出現してきました。特に、2022年9月頃からインドを中心に急速に流行を拡大したオミクロンXBB株は、遺伝子組換えにより生じた珍しいタイプの変異株です。本研究では、オミクロンXBB株の出現に至った進化的経路と、オミクロンXBB株のウイルス学的特性を明らかにするため、詳細な解析を行いました。
進化系統解析の結果、オミクロンXBB株は、2種類のオミクロン株(オミクロンBJ.1株とBM.1.1.1株[BA.2.75株の子孫株])がスパイクタンパク質のRBDにおいて遺伝子組換えを起こしたことで生じた変異株であることが明らかとなりました(図1)。
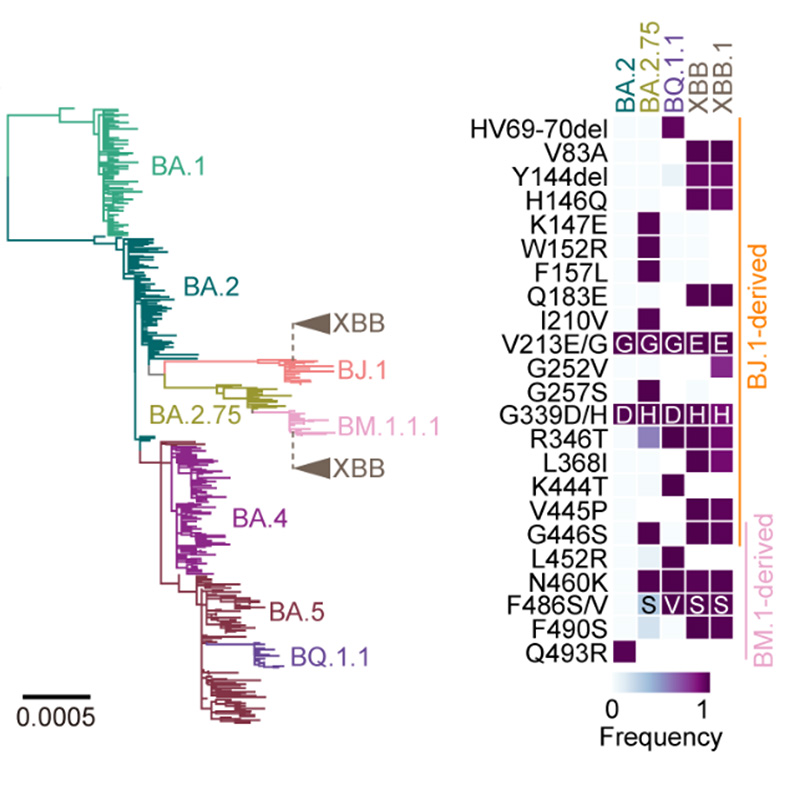
図1 2つのオミクロン亜株からXBBの出現
オミクロン亜株の系統樹。オミクロンBA.1株が出現した後、様々なオミクロン亜株が継続的に出現し、流行を引き起こしてきた。オミクロンBA.2株からBJ.1株とBM.1.1.1株が出現し、それらの遺伝子組み換え体としてXBB亜系統が出現した。
オミクロンXBB株の出現時期を推定したところ、2022年6月中旬から7月上旬であると推定されました。また、流行動態解析から、オミクロンXBB株の実効再生産数注8は、祖先株であるオミクロンBJ.1株とBM.1.1.1株よりも高いことがわかりました(図2)。この結果は、オミクロンXBB株の出現に際して起こった遺伝子組換えにより、ウイルスの実効再生産数が上昇したことを示唆しています。なお、SARS-CoV-2の進化史において、遺伝子組換えによる実効再生産数の明確な上昇が観察されたのは、オミクロンXBB株の例が初めてです。
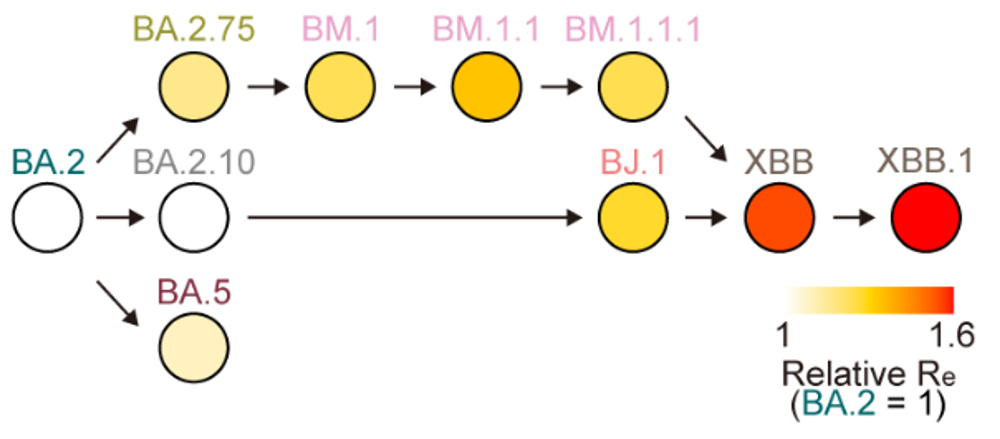
図2 遺伝子組換え体XBBのウイルス適応度
BA.2株を共通の祖先を持つBM.1.1とBJ.1がXBBに遺伝子組換えによって出現した際に、適応度が増大したことを示す。
流行動態解析の結果、2022年11月時点において、COVID-19流行の主流株がヨーロッパ・アメリカ・アフリカ地域と南・東南アジアおよびオセアニア地域の間で異なることが明らかになりました。すなわち、前者の国ではオミクロンBA.5株の子孫株であるオミクロンBQ.1.1株が、後者の国ではオミクロンXBB株が主流に流行していることがわかりました(図3a)。また、オミクロンXBB株の実効再生産数は、オミクロンBQ.1.1株の実効再生産数と同等に高いことが明らかになりました(図3b)。
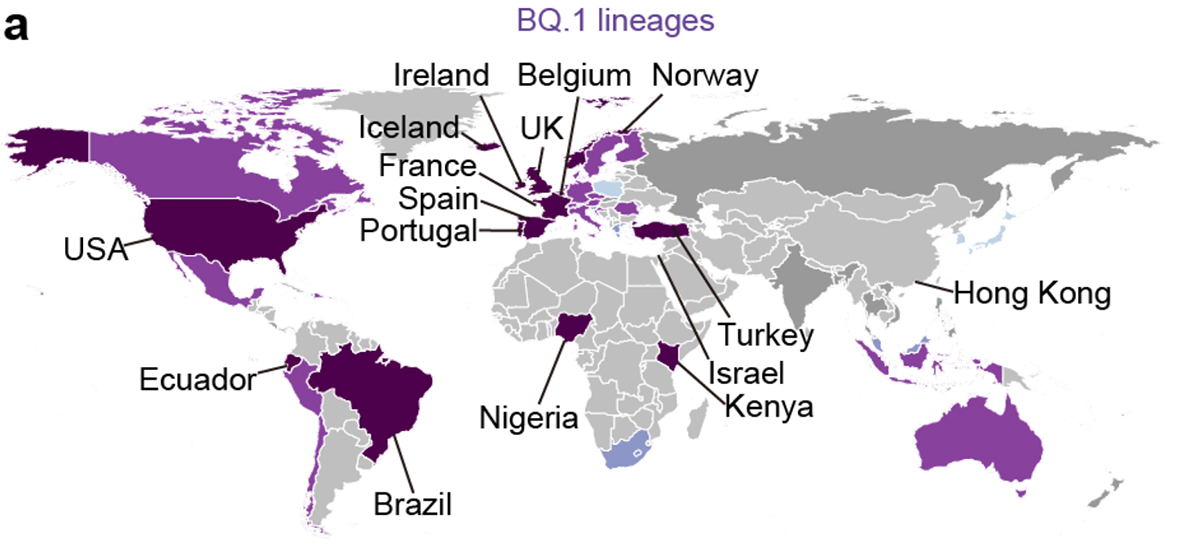
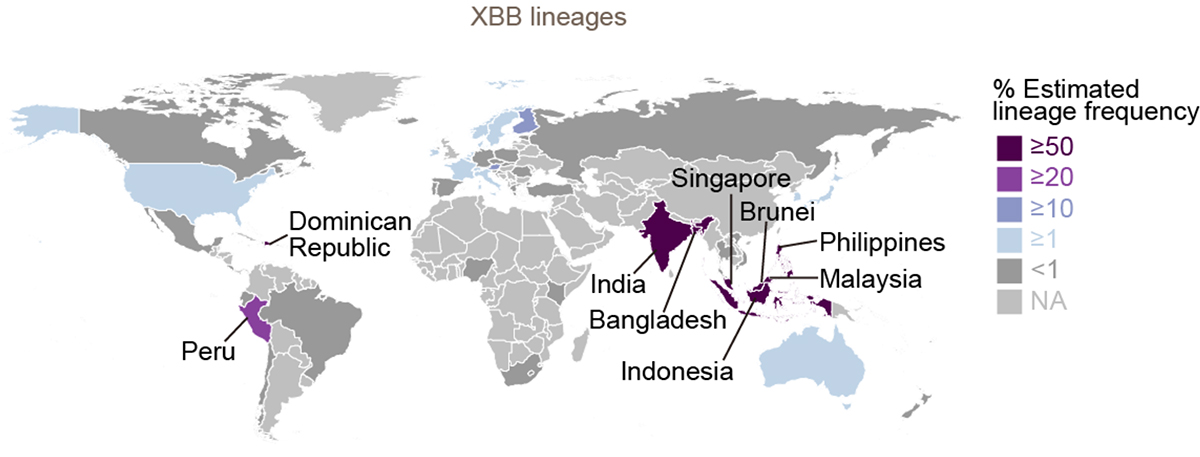
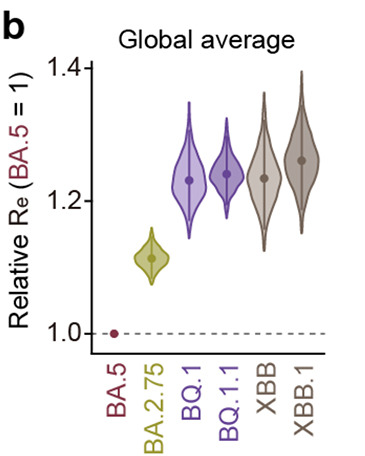
図3 オミクロンXBB株とオミクロンBQ.1.1株の流行
(a) 2022年11月15日時点におけるオミクロンXBB株とオミクロンBQ.1.1株の流行頻度。
(b) オミクロンXBB株とオミクロンBQ.1.1株の実効再生産数(Re)の比較。
オミクロンBA.5株の実効再生産数を1としたときの値を示している。
オミクロンXBB株についての詳細なウイルス性状解析実験を行いました。シュードウイルス注9を用いた中和試験注10の結果、XBB株は液性免疫からの逃避能を著しく上昇させた変異株であることが明らかになりました(図4a、4b)。この免疫逃避能の上昇は、スパイクタンパク質に存在する複数のアミノ酸置換の複合的な効果により得られたものであることがわかりました(図4a、4b)。これらアミノ酸置換は、祖先株であるBA.2.75株、およびBA.2.75株の祖先株であるBA.2株よりもACE2結合能と侵入効率が向上し、高い合胞体形成活性注11を有することがわかりました(図4c)。生体での呼吸器系の環境を模倣する気道チップを駆使した実験の結果、オミクロンXBB株は、オミクロンBA.2.75株よりも高い呼吸器上皮―血管内皮バリアの破壊能を持つことがわかりました(図4d)。さらに、ウイルス感染受容体であるACE2とのクライオ電子顕微鏡構造解析を行い、オミクロンXBB株のスパイクタンパク質がACE2に対し高い親和性を持つことの構造学的な基盤を同定しました。
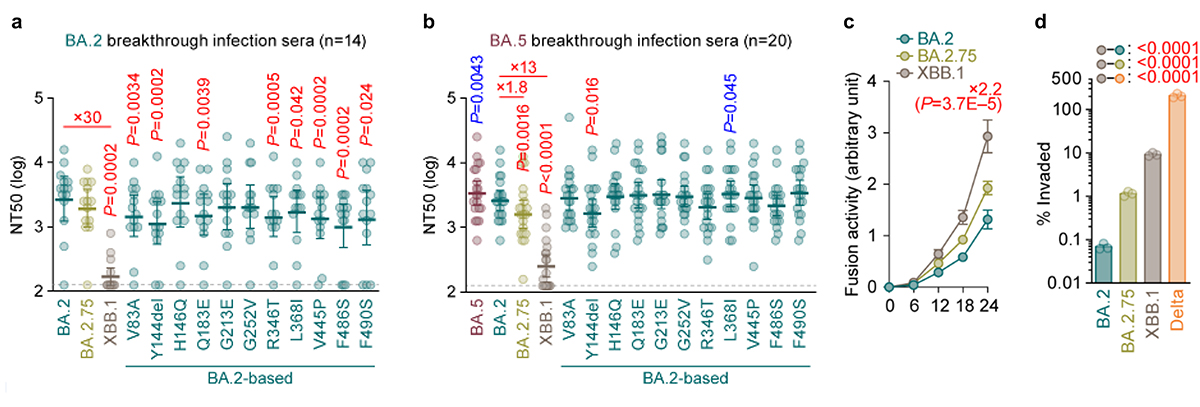
図4 オミクロンXBB.1株のウイルス学的特性
オミクロンBA.2株(a)とBA.5株(b)のブレークスルー感染者血清を用いたウイルス中和試験。中和力価の低いほどウイルスの中和抗体に対する逃避能が高いことを意味する。
(c) シュードウイルスを用いた合胞体形成活性の評価。
(d) 気道チップを用いたウイルスの呼吸器―血管内皮バリア破壊能の評価。
最後に、ハムスターモデルを用いた感染実験により、オミクロンXBB株の個体内感染動態と病原性を評価しました。その結果、オミクロンXBB株のハムスターの肺における増殖力は、祖先型であるオミクロンBA.2.75株よりもやや低下しており、また病原性もやや低いことが明らかとなりました(図5)。
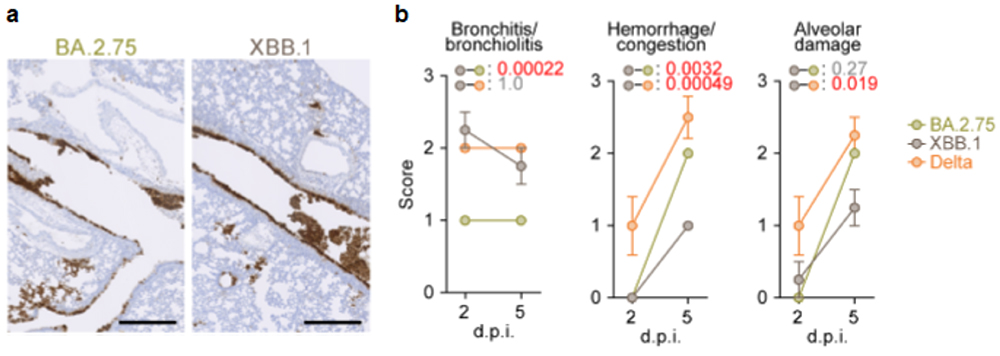
図5 オミクロンXBB.1株のハムスターモデルにおける増殖性と組織障害性
(a) 感染後2日目のハムスター肺におけるウイルスの増殖。ウイルスが感染している細胞
(Nタンパク質陽性細胞)は、茶色に免疫染色されている。
(b) ハムスター肺の組織病理学的障害スコア。値が高いほど組織障害の程度が強いことを示す。
本研究で解析したオミクロンXBB株は、その後さらに多様化し、様々な子孫株を生み出すに至りました。特に、2023年に入り、オミクロンXBB株よりも高い実効再生産数を示すオミクロンXBB.1.5株やXBB.1.16株などが相次いで出現し、急速に流行を拡大しました。2023年4月現在、このようなオミクロンXBB株の子孫株が全世界におけるCOVID-19流行の主流株となっています。
G2P-Japanでは、SARS-CoV-2の進化・流行動態を司る原理の解明に関する研究、および、出現が続くさまざまな変異株について、ウイルス学的な特性の解析や、中和抗体や治療薬への感受性の評価、病原性についての研究に取り組んでいます。G2P-Japanコンソーシアムでは、今後も、SARS-CoV-2の変異(Genotype)の早期捕捉と、その変異がヒトの免疫やウイルスの病原性・複製に与える影響(Phenotype)を明らかにするための研究を推進します。
本研究で解析したオミクロンXBB株は、その後さらに多様化し、様々な子孫株を生み出すに至りました。特に、2023年に入り、オミクロンXBB株よりも高い実効再生産数を示すオミクロンXBB.1.5株やXBB.1.16株などが相次いで出現し、急速に流行を拡大しました。2023年4月現在、このようなオミクロンXBB株の子孫株が全世界におけるCOVID-19流行の主流株となっています。
G2P-Japanでは、SARS-CoV-2の進化・流行動態を司る原理の解明に関する研究、および、出現が続くさまざまな変異株について、ウイルス学的な特性の解析や、中和抗体や治療薬への感受性の評価、病原性についての研究に取り組んでいます。G2P-Japanコンソーシアムでは、今後も、SARS-CoV-2の変異(Genotype)の早期捕捉と、その変異がヒトの免疫やウイルスの病原性・複製に与える影響(Phenotype)を明らかにするための研究を推進します。
- 論文名
Virological characteristics of the SARS-CoV-2 XBB variant derived from recombination of two Omicron subvariants - ジャーナル名
Nature Communications - 著者
Tomokazu Tamura1,2,*, Jumpei Ito3,*, Keiya Uriu3,4,*, Jiri Zahradnik5,6,*, Izumi Kida7,*, Yuki Anraku8,*, Hesham Nasser9,10,*, Maya Shofa11,12,*, Yoshitaka Oda13,*, Spyros Lytras14,*, Naganori Nao15,16, Yukari Itakura2,17, Sayaka Deguchi18, Rigel Suzuki1,2, Lei Wang13,19, MST Monira Begum8, Shunsuke Kita8, Hisano Yajima20, Jiei Sasaki20, Kaori Sasaki-Tabata21, Ryo Shimizu9, Masumi Tsuda13,19, Yusuke Kosugi3,4, Shigeru Fujita3,4, Lin Pan3,22, Daniel Sauter3,23, Kumiko Yoshimatsu24, Saori Suzuki1,2, Hiroyuki Asakura25, Mami Nagashima25, Kenji Sadamasu25, Kazuhisa Yoshimura25, Yuki Yamamoto26, Tetsuharu Nagamoto26, Gideon Schreiber5, Katsumi Maenaka2,8,27,28, The Genotype to Phenotype Japan (G2P-Japan) Consortium, Takao Hashiguchi20, Terumasa Ikeda9, Takasuke Fukuhara1,2,30,31, Akatsuki Saito11,12,32, Shinya Tanaka13,19**, Keita Matsuno2,7,16,33**, Kazuo Takayama18,30**,
Kei Sato3,4,22,29,34,35,36**
*:These authors contributed equally
**:責任著者 - 著者の所属機関
- Department of Microbiology and Immunology, Faculty of Medicine, Hokkaido University, Sapporo, Japan
- Institute for Vaccine Research and Development, HU-IVReD, Hokkaido University, Sapporo, Japan
- Division of Systems Virology, Department of Microbiology and Immunology, The Institute of Medical Science, The University of Tokyo, Tokyo, Japan
- Graduate School of Medicine, The University of Tokyo, Tokyo, Japan
- Department of Biomolecular Sciences, Weizmann Institute of Science, Rehovot, Israel
- First Medical Faculty at Biocev, Charles University, Vestec-Prague, Czechia
- Division of Risk Analysis and Management, International Institute for Zoonosis Control, Hokkaido University, Sapporo, Japan
- Laboratory of Biomolecular Science and Center for Research and Education on Drug Discovery, Faculty of Pharmaceutical Sciences, Hokkaido University, Sapporo, Japan
- Division of Molecular Virology and Genetics, Joint Research Center for Human Retrovirus infection, Kumamoto University, Kumamoto, Japan
- Department of Clinical Pathology, Faculty of Medicine, Suez Canal University, Ismailia, Egypt
- Department of Veterinary Science, Faculty of Agriculture, University of Miyazaki, Miyazaki, Japan
- Graduate School of Medicine and Veterinary Medicine, University of Miyazaki, Miyazaki, Japan
- Department of Cancer Pathology, Faculty of Medicine, Hokkaido University, Sapporo, Japan
- Medical Research Council-University of Glasgow Centre for Virus Research, Glasgow, UK
- Division of International Research Promotion, International Institute for Zoonosis Control, Hokkaido University, Sapporo, Japan
- One Health Research Center, Hokkaido University, Sapporo, Japan
- Division of Molecular Pathobiology, International Institute for Zoonosis Control, Hokkaido University, Sapporo, Japan
- Center for iPS Cell Research and Application (CiRA), Kyoto University, Kyoto, Japan
- Institute for Chemical Reaction Design and Discovery (WPI-ICReDD), Hokkaido University, Sapporo, Japan
- Laboratory of Medical Virology, Institute for Life and Medical Sciences, Kyoto University, Kyoto, Japan
- Department of Medicinal Sciences, Graduate School of Pharmaceutical Sciences, Kyushu University, Fukuoka, Japan
- Graduate School of Frontier Sciences, The University of Tokyo, Kashiwa, Japan
- Institute for Medical Virology and Epidemiology of Viral Diseases, University Hospital Tübingen, Tübingen, Germany
- Institute for Genetic Medicine, Hokkaido University, Sapporo, Japan
- Tokyo Metropolitan Institute of Public Health, Tokyo, Japan
- HiLung, Inc., Kyoto, Japan
- Global Station for Biosurfaces and Drug Discovery, Hokkaido University, Sapporo, Japan
- Division of Pathogen Structure, International Institute for Zoonosis Control, Hokkaido University, Sapporo, Japan
- CREST, Japan Science and Technology Agency, Kawaguchi, Japan
- AMED-CREST, Japan Agency for Medical Research and Development (AMED), Tokyo, Japan
- Laboratory of Virus Control, Research Institute for Microbial Diseases, Osaka University, Suita, Japan
- Center for Animal Disease Control, University of Miyazaki, Miyazaki, Japan
- International Collaboration Unit, International Institute for Zoonosis Control, Hokkaido University, Sapporo, Japan
- International Research Center for Infectious Diseases, The Institute of Medical Science, The University of Tokyo, Tokyo, Japan
- International Vaccine Design Center, The Institute of Medical Science, The University of Tokyo, Tokyo, Japan
- Collaboration Unit for Infection, Joint Research Center for Human Retrovirus infection, Kumamoto University, Kumamoto, Japan
本研究は、佐藤佳教授に対する日本医療研究開発機構(AMED)新興・再興感染症に対する革新的医薬品等開発推進研究事業(JP22fk0108146、JP21fk0108494、JP21fk0108425, JP21fk0108432)、AMED 先進的研究開発戦略センター(SCARDA)ワクチン開発のための世界トップレベル研究開発拠点の形成事業(UTOPIA、JP223fa627001)、AMED SCARDAワクチン・新規モダリティ研究開発事業(JP223fa727002)、科学技術振興機構(JST) CREST(JPMJCR20H4)、および、高山和雄講師に対する日本医療研究開発機構(AMED)新興・再興感染症に対する革新的医薬品等開発推進研究事業(JP21fk0108425、JP21fk0108432)、AMED-CREST(JP21gm1610005)などの支援の下で実施されました。
(注1)研究コンソーシアム「The Genotype to Phenotype Japan (G2P-Japan)」
東京大学医科学研究所 システムウイルス学分野の佐藤佳教授が主宰する研究コンソーシアム。日本国内の様々な専門性を持つ若手研究者が参画し、多角的アプローチからウイルスの性質の解明に取り組んでいる。現在では、イギリスを中心とした諸外国の研究チーム・コンソーシアムとの国際連携も進めている。
(注2)オミクロン株(B.1.1.529, BA系統)
新型コロナウイルスの流行拡大によって出現した、顕著な変異を有する「懸念すべき変異株(VOC:variant of concern)」のひとつ。オミクロンBA.1株、オミクロンBA.5株、オミクロンBQ.1.1株(注12)、オミクロンXBB株などが含まれる。現在、日本を含めた世界各国で大流行している変異株である。
(注3)スパイクタンパク質
新型コロナウイルスが細胞に感染する際に、新型コロナウイルスが細胞に結合するためのタンパク質。現在使用されているワクチンの標的である。
(注4)受容体結合部位(RBD)
上述のスパイクタンパク質のうち、細胞の受容体に結合する領域(Receptor Binding Domain)。
(注5)遺伝子組換え
複数のウイルスの間で、遺伝子の一部が組み換わること。新型コロナウイルスの場合、異なる系統のウイルスが宿主に同時感染することで生じると考えられる。
(注6)液性免疫
B細胞が主体となって、抗体を作ることで異物に対抗する免疫。
(注7)ACE2
Angiotensin-Converting Enzyme 2(アンジオテンシン変換酵素2)の略称で、新型コロナウイルスが細胞に感染する際に受容体として機能する。
(注8)実効再生産数
特定の条件下において、1人のウイルス感染者が平均して何人の二次感染者を生み出せるかを表す尺度。本研究では変異株の流行拡大能力を比較する目的で使用している。
(注9)シュードウイルス
新型コロナウイルスのスパイクタンパク質を持たせたウイルス様粒子。細胞に一度感染できるが、増殖することはできない。
(注10)中和試験
抗体や抗体を含む血清がウイルスを不活化(中和)する能力を定量するための実験。ウイルスを中和する能力のある抗体は中和抗体と呼ばれる。新型コロナウイルスの場合、中和抗体はスパイクタンパク質に結合することでスパイクタンパク質と感染受容体であるACE2との相互作用を抑制する場合が多い。
(注11)合胞体形成活性
合胞体とは、新型コロナウイルスに感染した細胞が、スパイクタンパク質を細胞表面に発現し、周囲の細胞と融合することによって形成される大きな細胞塊のこと。合胞体形成活性とは、新型コロナウイルスのスパイクタンパク質を介して、合胞体を形成する能力のこと。
(注12)BQ.1.1
本研究成果とほぼ同時期にオミクロンBQ.1.1株に関する解析を実施し、下記論文を公表しています。
- 論文名
Convergent evolution of SARS-CoV-2 Omicron subvariants leading to the emergence of BQ.1.1 variant - ジャーナル名
Nature Communications - 著者
伊東潤平#, 鈴木理滋#, 瓜生慧也#, 板倉友香里#, Jiri Zahradnik#, 木村香菜子#, 出口清香#, 王磊#, Spyros Lytras#, 田村友和, 紀田泉, Hesham Nasser, Maya Shofa, MST Monira Begum, 津田真寿美, 小田義崇, 鈴木干城, 佐々木慈英, 田畑香織, 藤田滋, 吉松組子, 伊藤駿, 直亨則, 浅倉弘幸, 長島真美, 貞升健志, 吉村和久, 山本佑樹, 永元哲治, 倉持仁, Gideon Schreiber, The Genotype to Phenotype Japan (G2P-Japan) Consortium, 齊藤暁, 松野啓太, 高山和雄, 橋口隆生*, 田中伸哉*, 福原崇介*, 池田輝政*, 佐藤佳*
#:Equal contribution; *:Corresponding author