ニュース・イベント
News & Events
ニュース・イベント
News & Events
ニュース
News
2016年12月27日
Nat1はマウスES細胞の分化を誘導する特定のタンパク質の翻訳を促進する
ポイント
- マウスES細胞のNat1遺伝子を欠損させると、ground state注1)に類似した性質へと変化する。
- NAT1タンパク質は一般的な翻訳注2)開始因子eIF4G1とは異なるCap注3)非依存的な翻訳開始複合体を形成する。
- Nat1遺伝子はMap3k3の翻訳量を促進することで、分化を誘導している。
杉山逸未研究員、山中伸弥教授(CiRA未来生命科学開拓部門)らの研究グループは、Nat1というタンパク質がマウスES細胞において、分化を誘導するタンパク質の翻訳を促進することを明らかにしました。
Nat1は山中教授らが20年前に同定し、マウス発生の初期段階やES細胞の分化能に必須の遺伝子であることを報告していました。今回の論文では、NAT1は一般的な翻訳開始因子のeIF4G1と配列が似ていますが、eIF4G1とは異なるメカニズムでタンパク質翻訳を行っていることを明らかにしました。さらに、NAT1がMap3k3など分化誘導に必須なタンパク質の翻訳量を調整することにより、ES細胞の分化能に関っていることを明らかにしました。
この研究成果は2016年12月22日に米国科学誌「Proceedings of the National Academy of Sciences」に掲載されました。
NAT1 (Novel APOBEC1 target 1、別名:eIF4G2、p97、またはDAP5) は山中教授が1997年に、RNA編集酵素注4)であるAPOBEC1のターゲットとして世界に先駆けて同定した遺伝子です。NAT1は様々な遺伝子の翻訳開始に関わる因子eIF4G1のC末端側注5)アミノ酸配列の2/3と相同性を持つ構造を持ち、哺乳類細胞において恒常的に発現しています。山中教授らはこれまでに、Nat1はマウス胚発生の初期段階で必須な遺伝子であることを報告しています。またNat1を欠損させたマウスES細胞のコロニー(集合体)は、通常の扁平な形態から、ドーム状形態へと変化し、分化刺激に対して抵抗性を示すことも報告しています。この特徴は、ES細胞において分化シグナルであるERK及びGSK3を阻害することにより誘導される、より未分化な細胞が均一に存在する基底状態 (ground state)のES細胞 と類似していていました。しかしNat1欠損によってES細胞の性質が本当にground stateへ変化しているのか、また、Nat1による制御機構については明らかになっていませんでした。
本研究では、Nat1欠損により、マウスES細胞が、分化シグナルであるERK及びGSK3の阻害による、ES細胞のより未分化なground stateと同様の性質を有しているのか詳細に調べるとともに、ES細胞におけるNat1の分子制御機構を明らかにすることを目的としました。
1. Nat1欠損させたES細胞は、ground stateと類似する性質をもつ。
マウスの野生型注6)ES細胞は、分化シグナルであるERK及びGSK3の阻害剤 (2i) で処理した場合(2i+LIF)には、より多能性が高く均一なドーム状の形態の細胞集団(ground state)へと変化することが知られています。また、阻害剤を使わず標準的な方法で培養した場合(LIF)でもNat1を欠損させたES細胞のコロニーは、野生型ES細胞のground stateと同様の形態へと変化します (図1.A) 。
野生型ES細胞のground stateとNat1欠損ES細胞とで遺伝子発現解析を行いました(図1.B)。その結果、Nat1欠損ES細胞も、多能性関連遺伝子(Nanog, Pou5f1, Sox2, Esrrb, Klf4, Klf5, Tbx3)が野生型ES細胞のground stateと同じように高く発現していることが明らかとなりました。また、細胞内シグナル伝達経路への影響を調べたところ、野生型ES細胞のground stateと同様に、Nat1欠損ES細胞ではERKのリン酸化が抑制され、また未分化な状態を促進するシグナルのSTAT3のリン酸化が活性化していることがわかりました。その一方で、ERK/GSK3阻害剤(2i)で処理した場合とは異なり、GSK3のリン酸化は抑制されていませんでした(図1.C)。これらの結果から、Nat1を欠損させることにより、そのES細胞の性質をground stateと同一ではありませんが、類似した性質へと遷移させることが明らかになりました。
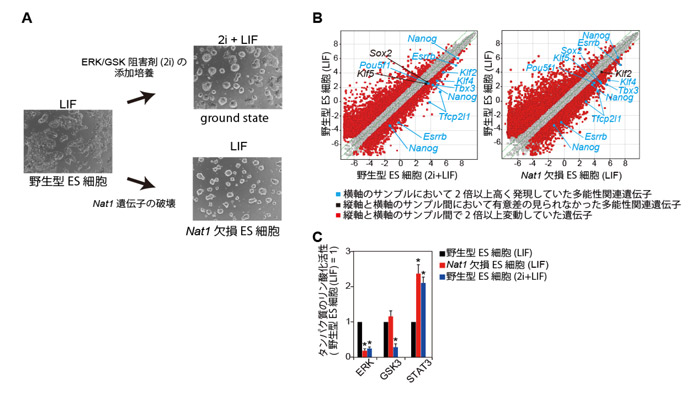
図1. Nat1欠損ES細胞と野生型ES細胞のground stateにおける (A) ES細胞のコロニーの形態と、(B) 遺伝子発現比較 (C) 細胞内シグナル伝達経路のタンパク質のリン酸化活性の定量比較。
(B) 2サンプル間の遺伝子発現の散布図。四角形の点は縦軸と横軸の2サンプル間の遺伝子の発現量の比を示しています。四角形の点が、右上から左下を結ぶ対角線から離れそれぞれの軸に近づくほど、それぞれの軸でその遺伝子が高く発現していることを表しています。
NAT1の機能を明らかにするために免疫沈降法注7)と質量分析法注8)を用いて、NAT1に結合するタンパク質を同定しました。NAT1は翻訳開始因子eIF3やリボゾーム注9)タンパク質と結合していました。これはNAT1とC末端側アミノ酸配列の2/3が類似しているeIF4G1と共通していました。しかし、eIF4G1とは異なり、NAT1は、mRNAの5'末端であるCap構造を認識するeIF4Eには結合していませんでした。一方で、NAT1は、RNA結合タンパク質であるFXRやPRRC2と強く結合していました。つまりNAT1は、Cap構造依存的に翻訳を開始するeIF4G1複合体とは異なり、Cap構造に依存しない翻訳開始に関与していると考えられました(図.2)。
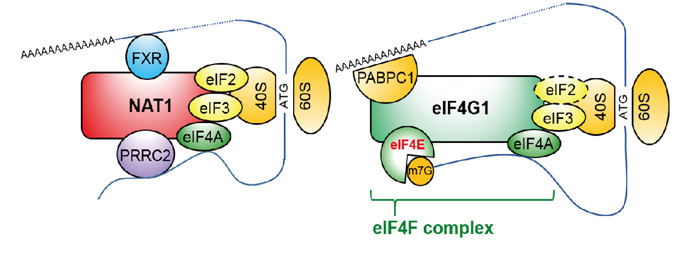
図.2 NAT1によるCap非依存的な翻訳開始複合体(左)と、eIF4G1によるCap依存的な翻訳開始複合体(右)のモデル図
Nat1が翻訳に関与しているmRNAの種類を調べるため、リボゾームプロファイリングをという手法を用いて、Nat1欠損により翻訳量が変化しているRNAを同定しました。その結果、多能性幹細胞の分化を促進するERKを制御しているMap3k3遺伝子の翻訳量がNat1によって促進されていました (図.3)。つまりNat1欠損ES細胞ではMap3k3の翻訳が抑制されることで、下流の分化シグナルが抑制され、未分化な状態を保っていると考えられました。
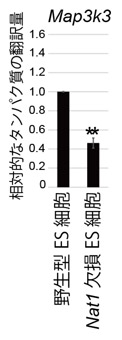
図.3 Nat1はMap3k3の翻訳を促進している。
Map3k3の翻訳が抑制されることで、Nat1欠損ES細胞がground stateに類似した性質を獲得していることを明らかにするために、Nat1欠損ES細胞にMap3k3を強制発現させたES細胞株を樹立しました。Map3k3を強制発現させたNat1欠損ES細胞では、コロニーの形態が未分化様のドーム状の形態から野生型ES細胞に似た扁平な形態へと変化しました (図4.A)。またERKのリン酸化は活性化(図4.B)されており、主要な多能性関連遺伝子の発現量は抑制(図4.C)されていました。また分化マーカーの発現量は増加(図4.D)していました。これらの結果から、Map3k3はground stateに類似した細胞から分化を誘導している可能性が示唆されました。
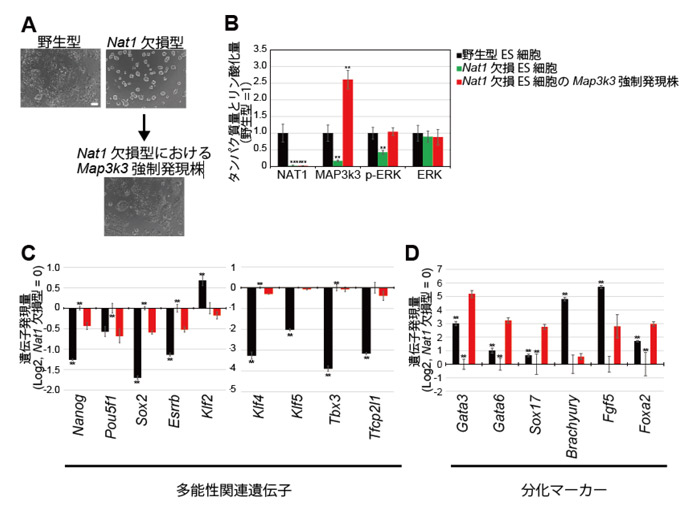
図.4 Map3k3を強制発現させたNat1欠損ES細胞の性質の変化
(A) Nat1欠損ES細胞にMap3k3を強制発現させたコロニーの形態変化。
(B) リン酸化ERK (p-ERK)の活性の変化。
(C) 多能性関連遺伝子の発現量の変化。
(D) 分化マーカーの発現量の変化。
これらの結果から、マウスES細胞は、Nat1を欠損させることで、ERK/GSK3阻害剤で処理した場合のground stateと同一ではないが、類似した状態へと変化することを明らかとしました。その分子制御機構の一つとして、NAT1は、eIF4G1複合体とは異なり、Cap非依存的な翻訳開始因子複合体を形成し、Map3k3の翻訳を促進していることが示され、またMap3k3はES細胞を分化させる性質をもつ遺伝子であることがわかりました。 本研究により、Nat1による翻訳制御を介したマウスES細胞の多能性の制御機構が解明されました。これらの研究成果は、ヒト多能性幹細胞(iPS/ES細胞)をより安定的で多能性の高い基底状態へ性質を変化させるための研究への応用が期待されます。
- 論文名
Nat1 promotes translation of specific proteins that induce differentiation of mouse embryonic stem cells - ジャーナル名
Proceedings of the National Academy of Sciences -
著者
Hayami Sugiyama1, Kazutoshi Takahashi1,2, Takuya Yamamoto3,4, Mio Iwasaki1, Megumi Narita1, Masahiro Nakamura1, Tim A. Rand2, Masato Nakagawa1, Akira Watanabe1,3,4, and Shinya Yamanaka1,2*
*責任著者
- 著者の所属機関
- 京都大学iPS細胞研究所(CiRA)
- グラッドストーン研究所
- 京都大学物質--細胞システム統合拠点(iCeMS)
- 科学技術振興機構 CREST
本研究は、下記機関より資金的支援を受けて実施されました。
- AMED 再生医療実現拠点ネットワークプログラム(iPS細胞研究中核拠点)
- iPS細胞研究基金
注1) 基底状態 (ground state)のES細胞
分化シグナルであるERK及びGSK3の阻害剤を添加した培地で培養したマウスの野生型ES細胞の性質。標準的なマウスES細胞培養培地で培養した場合に比べ、多能性がより均一かつ高く、また性質が安定した状態。
注2) 翻訳
細胞内の遺伝子情報はDNAからmRNAに転写されることで核の外に持ち出される。mRNAの配列情報をリボソームが読み取り、対応したアミノ酸をつなぎ合わせることでタンパク質が作られる。mRNAからタンパク質へと情報を移すことを翻訳と言う。
注3)Cap
細胞質で翻訳されるmRNAの先頭側(5'末端側)に一般的に存在する構造。mRNAの安定性や翻訳開始などに関与していることがわかっている。
注4)RNA編集酵素
mRNAを編集する酵素。一部の塩基を変換する反応を触媒する。
注5)C末端側
タンパク質はアミノ酸が数珠つなぎのように多数連なっている。アミノ酸の中にあるアミノ基(-NH2)とカルボキシル基(-COOH)が反応して隣のアミノ酸と結合する。この時タンパク質の一番端にはそれぞれ1個ずつのアミノ基とカルボキシル基が残る。このアミノ基側をN末端、カルボキシル基側をC末端と言う。通常タンパク質合成はN末端側から始まり、C末端側にアミノ酸を連ねていく。
注6) 野生型
遺伝子の欠損や変異のない、通常の一般的なタイプ。
注7)免疫沈降法
特定のタンパク質と結合する抗体を使って、そのタンパク質が含まれる複合体だけを沈降させて分離する方法。この時に目的としたタンパク質と複合体を作りやすい他のタンパク質などが同時に検出される。
注8)質量分析法
物質の重さ(質量)を分析する方法。すでに知られているタンパク質などであれば、質量分析法で解析することでどの様なタンパク質が含まれているのか同定することができる。
注9)リボゾーム
mRNAの情報を読み取ってタンパク質へと翻訳する場。マウスやヒトでは40Sと60Sという2つのサブユニットからなる。