ニュース・イベント
News & Events
ニュース・イベント
News & Events
ニュース
News
2024年6月6日
SARS-CoV-2関連コロナウイルスBANAL-20-236株のウイルス学的特性の解析
ポイント
- 新型コロナウイルス(SARS-CoV-2)に近縁なコロナウイルスであるBANAL-20-236株(B236株)は、東南アジアにてコウモリから分離されたウイルスである。
- 本研究は、B236株の培養細胞やヒトオルガノイドにおける感染性、ハムスターにおける病原性をSARS-CoV-2やその変異体と比較しつつ、明らかにした。
- B236株は、SARS-CoV-2と比較して、ヒト気道上皮細胞では増殖能が低い一方で、ヒト大腸オルガノイドでは増殖能が高いという組織指向性があることが示された。
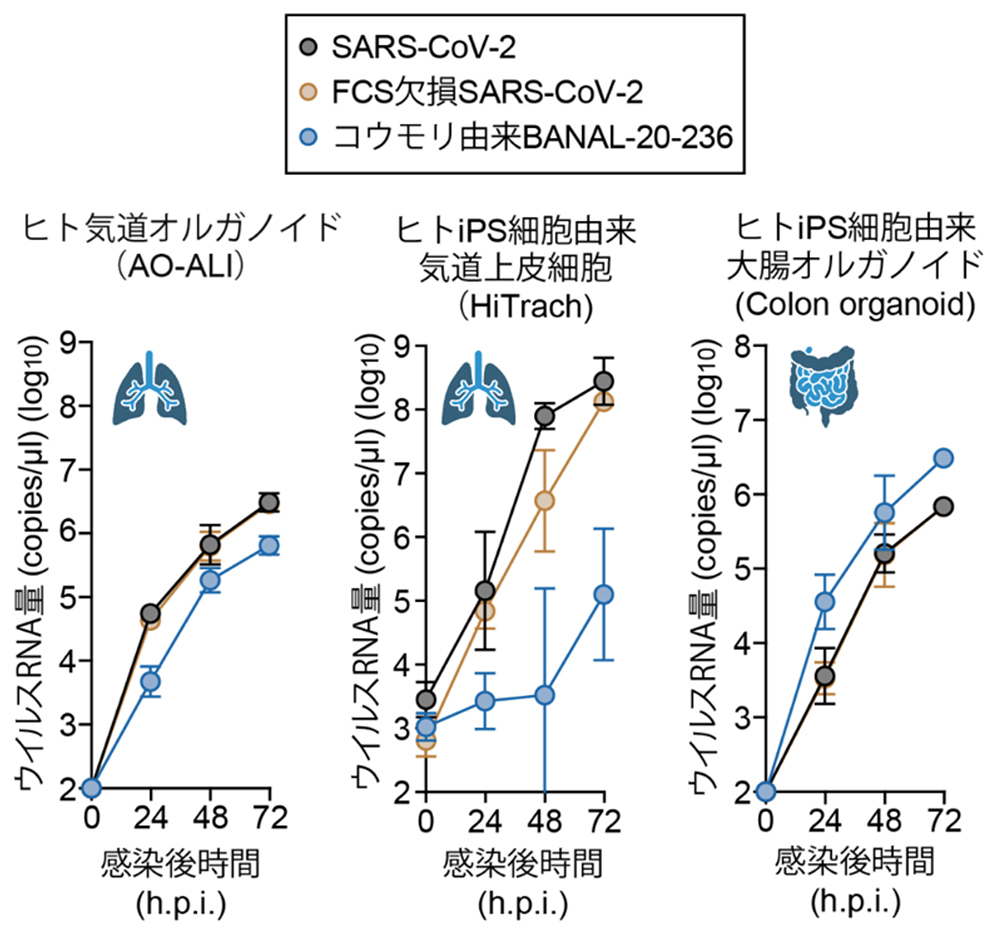
図:B236株は、SARS-CoV-2と異なる組織指向性を示す
高山和雄 講師(CiRA増殖分化機構研究部門)、佐藤佳教授(東京大学医科学研究所)、研究コンソーシアム「The Genotype to Phenotype Japan(G2P-Japan)」注1)らは、SARS-CoV-2に近縁なコウモリ由来のウイルスであるBANAL-20-236株(B236株)のウイルス学的特性を、ヒト気道上皮細胞や大腸オルガノイド、ハムスターモデル等を用いて解析しました。
ヒト呼吸器細胞モデルや大腸オルガノイドでの細胞実験により、B236株はSARS-CoV-2と比較して、ヒト気道上皮細胞よりも大腸オルガノイドへの指向性を持つことが示されました。また、ハムスターモデルを用いた感染実験では、B236株はSARS-CoV-2に比べて病原性が低いことが示されました。
本研究成果は2024年6月4日、英国科学雑誌「eBioMedicine」オンライン版で公開されました。
近年、SARS-CoV-2に近縁なウイルス(SARS-CoV-2関連コロナウイルス)注2)が、コウモリやセンザンコウ等の野生動物から多数発見されています。この事実から、SARS-CoV-2の祖先のウイルスも、元々はコウモリが保有していたウイルスであり、それがヒトに異種間伝播注3)したのではないかと考えられています。しかしながら、多数発見されているSARS-CoV-2関連コロナウイルスの中で、どのような特徴を持つウイルスがヒトに異種間伝播し、パンデミックを起こしうるのかについてのメカニズムには不明な点が多くあります。
BANAL-20-236株(B236株)は、2022年にラオスにてキクガシラコウモリ科のコウモリから分離されたSARS-CoV-2関連コロナウイルスで、実験的にヒト細胞に感染可能なことが報告されています。本研究では、コウモリ由来のB236株とSARS-CoV-2のウイルス学的特性を比較することで、パンデミックを起こすウイルスの特徴について理解することを目指しました。
B236株とSARS-CoV-2の大きな違いとして、ウイルスのスパイク(S)タンパク質注4)のFurin開裂部位(FCS)の有無が挙げられます。B236株を含む野生動物由来のSARS-CoV-2関連コロナウイルスにはFCSが存在しませんが、SARS-CoV-2にはFCSが存在します。SARS-CoV-2においては、FCSが動物モデルでのウイルスの病原性や伝播力に重要であることが先行研究で報告されています。我々は、SARS-CoV-2関連コロナウイルスにおいてFCSが与える影響について理解するため、B236株とSARS-CoV-2の従来株(WK-521株)に加えて、人工的に作製したSARS-CoV-2のFCSを欠損させたウイルス(SC2ΔFCS)を用意し、ヒトオルガノイドやハムスターモデルを使って、これらのウイルスの特性を比較しました(図1)。
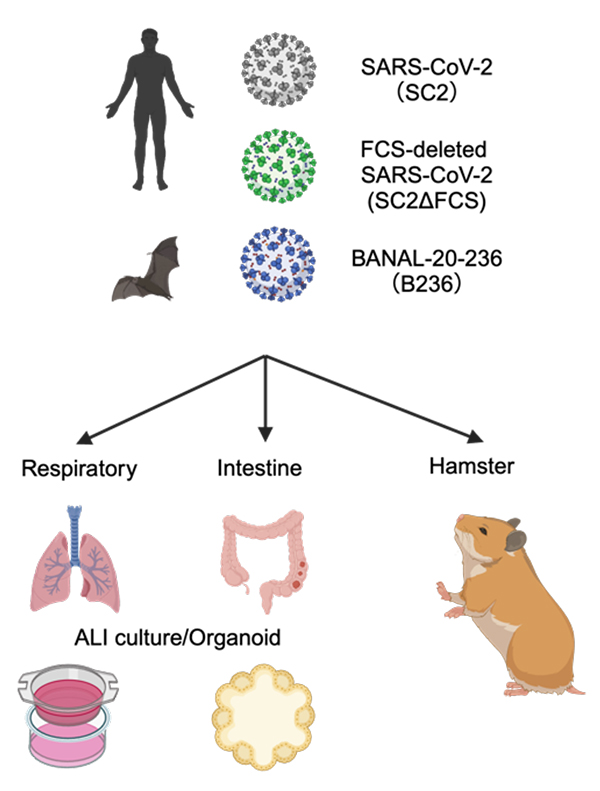
図1. 本研究で使用したウイルスと実験モデルの概要
BANAL-20-236株(B236株)、SARS-CoV-2(SC2)、FCS欠損SARS-CoV-2(SC2ΔFCS)のウイルス学的特性をヒトオルガノイドやハムスターモデルを用いて解析した。
まず、ヒト細胞モデルを用いて上記3つのウイルスの増殖能を比較しました。ヒト呼吸器細胞、特にヒト人工多能性幹細胞(iPSC)注5)由来の気道上皮細胞(HiTrach)では、B236株はSARS-CoV-2やSC2ΔFCSよりも顕著に増殖能が低いことが分かりました。一方、ヒトiPSC由来の大腸オルガノイドでは、B236株の方がSARS-CoV-2やSC2ΔFCSよりも高い増殖能を示すことが分かりました(図2)。
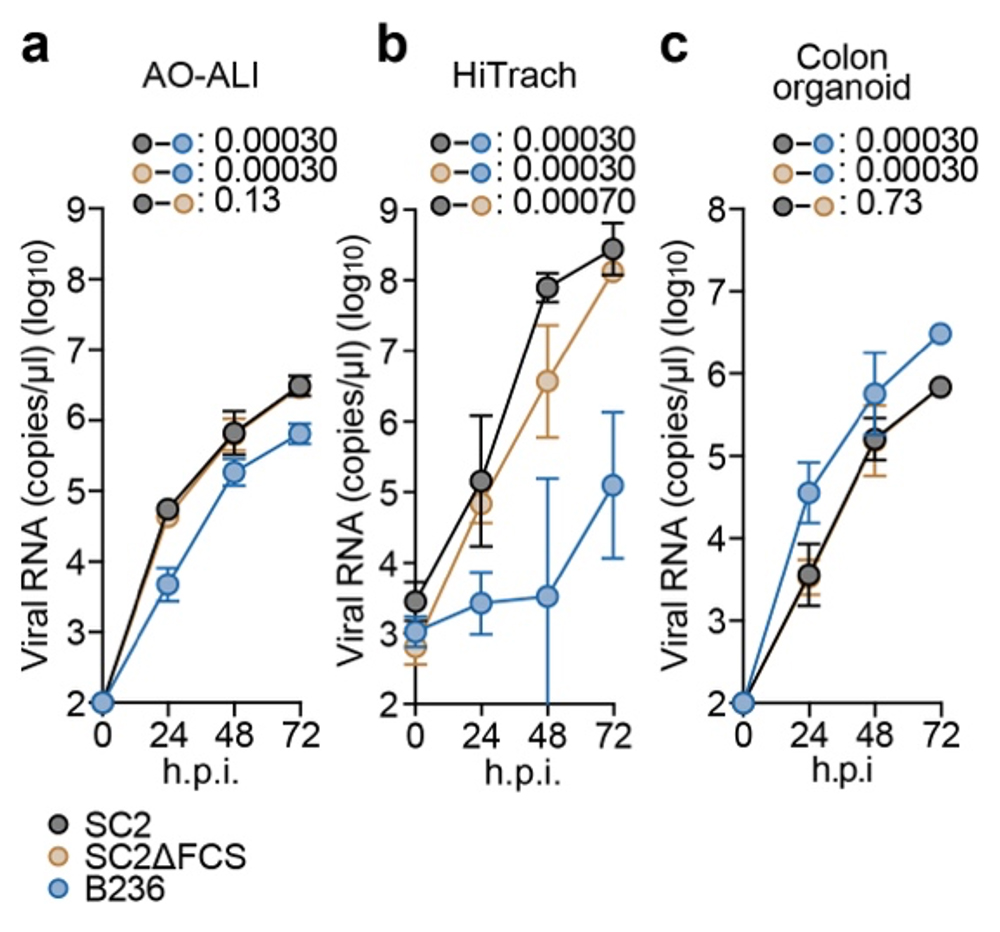
図2. 細胞株およびヒトオルガノイドでのウイルス感染実験
多種の細胞を用いて、BANAL-20-236株(B236株)、SARS-CoV-2(SC2)、FCS欠損SARS-CoV-2(SC2ΔFCS)の増殖能を比較している。a)気液界面(ALI)培養したヒト気道オルガノイド、b)ヒトiPSC由来の気道上皮細胞、c) ヒトiPSC由来の大腸オルガノイドを実験に使用した。統計検定は、時系列データの重回帰解析により実施し、Holm法で算出したfamilywise error ratesを図中に示している。
次に、これらのウイルスの持つ合胞体形成活性注6)を測定しました。Sタンパク質発現細胞を用いた細胞融合試験と、実際のウイルスを用いたプラークアッセイにより、B236株は、SARS-CoV-2と比較して、低い合胞体形成活性を持つことが分かりました(図3)。
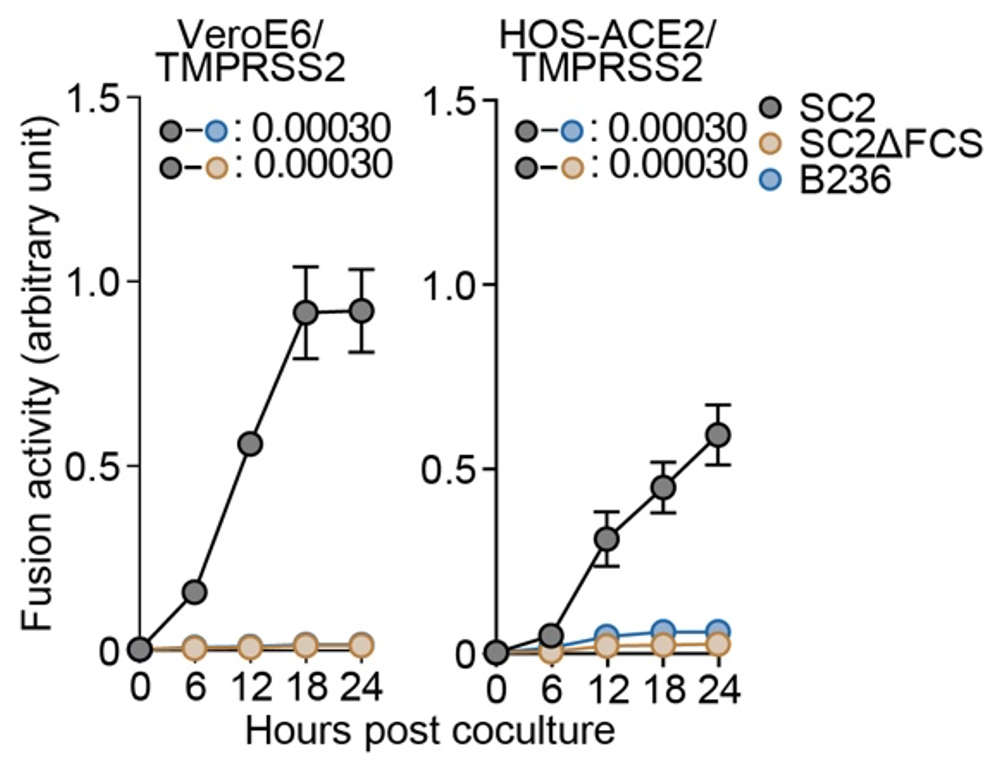
図3. 合胞体形成活性の評価
スパイクタンパク質発現細胞を用いた細胞融合試験。標的細胞には、VeroE6/TMPRSS2細胞とHOS-ACE2/TMPRSS2細胞を使用した。統計検定は、時系列データの重回帰解析により実施し、Holm法で算出したfamilywise error ratesを図中に示している。
最後に、B236株の病原性について解析するため、ハムスターモデルを用いた感染実験を実施しました。SARS-CoV-2感染ハムスターが明らかな体重減少、肺機能の低下を示すのに対し、SC2ΔFCS感染ハムスターはわずかな体重減少、B236株感染ハムスターに関しては、ほぼ非感染のコントロール群と変わらない結果となりました(図4)。先行研究でも示されている通り、FCSの欠損はSARS-CoV-2の病原性の低下に関与していますが、B236株感染ハムスターはSC2ΔFCS感染ハムスター以上に病原性が低いことから、B236株にはFCSの欠損以外にも、病原性の低下に関わる因子があることが示唆されます。
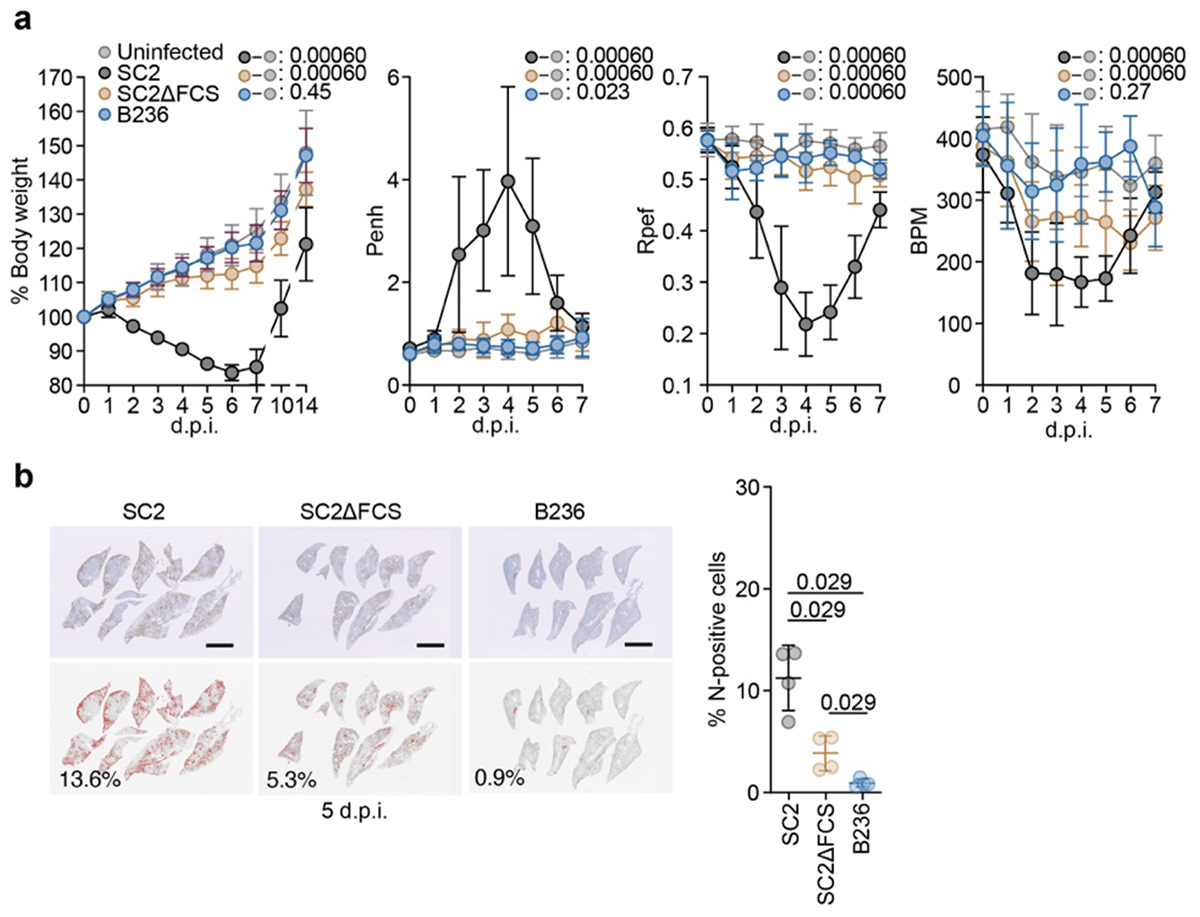
図4.ハムスターモデルを用いた病原性解析
a) ウイルス感染ハムスターの体重の推移と肺機能の評価, b) 感染ハムスター肺での、ウイルスNタンパク質の免疫染色像および肺中のNタンパク質陽性細胞の割合。統計検定は、a)では時系列データの重回帰解析により実施し、Holm法で算出したfamilywise error ratesを図中に示している。b)は、Mann-WhitneyのU検定より算出したP値を示している。
本研究では、B236株はSARS-CoV-2に比べて、合胞体形成活性や病原性が低いこと、また、ヒト気道上皮細胞よりも大腸オルガノイドへの指向性を持つことが示されました。B236株を含む多くのコウモリのSARS-CoV-2関連コロナウイルスは、コウモリの糞便または肛門スワブから検出されているため、今回の結果は、コウモリ由来のウイルスが呼吸器よりも腸管で主に複製されるのではないかという仮説を支持しています。我々は、今後も野生動物由来のコロナウイルスの特徴を明らかにするとともに、パンデミックを起こしたウイルスとの比較を通じて、ウイルスの異種間伝播のメカニズムを明らかにするための研究を推進します。
※一部の図は、Biorender.comを使用して作成しました。
- 論文名
Virological characteristics of a SARS-CoV-2-related bat coronavirus, BANAL-20-236 - ジャーナル名
eBiomMedicine - 著者
Shigeru Fujita1,2**, Arnon Plianchaisuk1**, Sayaka Deguchi3**, Hayato Ito4**, Naganori Nao5,6,7**, Lei Wang8,9**, Hesham Nasser10,11, Tomokazu Tamura4,5,6, Izumi Kimura1, Yukie Kashima12, Rigel Suzuki4,6, Saori Suzuki4,6, Izumi Kida13, Masumi Tsuda8,9, Yoshitaka Oda8, Rina Hashimoto3, Yukio Watanabe3, Keiya Uriu1,2, Daichi Yamasoba1,14, Ziyi Guo1, Alfredo A. Hinay Jr.1, Yusuke Kosugi1,2, Luo Chen1,12, Lin Pan1,12, Yu Kaku1, Hin Chu15, Flora Donati16,17, Sarah Temmam18,19, Marc Eloit18,19, Yuki Yamamoto20, Tetsuharu Nagamoto20, Hiroyuki Asakura21, Mami Nagashima21, Kenji Sadamasu21, Kazuhisa Yoshimura21, Yutaka Suzuki12, The Genotype to Phenotype Japan (G2P-Japan) Consortium, Jumpei Ito1,22, Terumasa Ikeda10, Shinya Tanaka8,9*, Keita Matsuno5,6,13,23*, Takasuke Fukuhara4,5,6,24*, Kazuo Takayama3*, Kei Sato1,2,12,22,25,26,27*
**:筆頭著者、*:責任著者 - 著者の所属機関
- Division of Systems Virology, Department of Microbiology and Immunology, The Institute of Medical Science, The University of Tokyo, Tokyo, Japan
- Graduate School of Medicine, The University of Tokyo, Tokyo, Japan
- Center for iPS Cell Research and Application (CiRA), Kyoto University, Kyoto, Japan
- Department of Microbiology and Immunology, Faculty of Medicine, Hokkaido University, Sapporo, Japan
- One Health Research Center, Hokkaido University, Sapporo, Japan
- Institute for Vaccine Research and Development (IVReD), Hokkaido University, Sapporo, Japan
- Division of International Research Promotion, International Institute for Zoonosis Control, Hokkaido University, Sapporo, Japan
- Department of Cancer Pathology, Faculty of Medicine, Hokkaido University, Sapporo, Japan
- Institute for Chemical Reaction Design and Discovery (WPI-ICReDD), Hokkaido University, Sapporo, Japan
- Division of Molecular Virology and Genetics, Joint Research Center for Human Retrovirus infection, Kumamoto University, Kumamoto, Japan
- Department of Clinical Pathology, Faculty of Medicine, Suez Canal University, Ismailia, Egypt
- Graduate School of Frontier Sciences, The University of Tokyo, Chiba, Japan
- Division of Risk Analysis and Management, International Institute for Zoonosis Control, Hokkaido University, Sapporo, Japan
- Faculty of Medicine, Kobe University, Kobe, Japan
- State Key Laboratory of Emerging Infectious Diseases, Department of Microbiology, School of Clinical Medicine, Li Ka Shing Faculty of Medicine, The University of Hong Kong, Pokfulam, Hong Kong Special Administrative Region, China
- Institut Pasteur, Université Paris Cité, CNRS UMR 3569, Molecular Genetics of RNA Viruses Unit, Paris, France
- Institut Pasteur, Université Paris Cité, National Reference Center for Respiratory Viruses, Paris, France
- Institut Pasteur, Université Paris Cité, Pathogen Discovery Laboratory, Paris, France
- Institut Pasteur, Université Paris Cité, The WOAH(OIE) Collaborating Center for the Detection and Identification in Humans of Emerging Animal Pathogens, Paris, France
- HiLung Inc., Kyoto, Japan
- Tokyo Metropolitan Institute of Public Health, Tokyo, Japan
- International Research Center for Infectious Diseases, The Institute of Medical Science, The University of Tokyo, Tokyo, Japan
- International Collaboration Unit, International Institute for Zoonosis Control, Hokkaido University, Sapporo, Japan
- Laboratory of Virus Control, Research Institute for Microbial Diseases, Osaka University, Suita, Japan
- International Vaccine Design Center, The Institute of Medical Science, The University of Tokyo, Tokyo, Japan
- Collaboration Unit for Infection, Joint Research Center for Human Retrovirus infection, Kumamoto University, Kumamoto, Japan
- MRC-University of Glasgow Centre for Virus Research, Glasgow, UK
本研究は、佐藤佳教授に対する日本医療研究開発機構(AMED)「先端国際共同研究推進プログラム(ASPIRE)(JP23jf0126002)」、AMED 「新興・再興感染症に対する革新的医薬品等開発推進研究事業(JP243fa727002)」、AMED 先進的研究開発戦略センター(SCARDA)「ワクチン開発のための世界トップレベル研究開発拠点の形成事業(UTOPIA, JP243fa627001h0003)、AMED SCARDA「ワクチン・新規モダリティ研究開発事業(JP243fa727002)」、科学技術振興機構(JST) CREST(JPMJCR20H4)、日本学術振興会(JSPS)「国際共同研究加速基金(国際先導研究)(JP23K20041)」、JSPS 「基盤研究(A)(JP24H00607)」、伊東 潤平准教授に対するJST「さきがけ(JPMJPR22R1)」、JSPS 「若手研究(JP23K14526)」などの支援の下で実施されました。
注1)研究コンソーシアム「The Genotype to Phenotype Japan (G2P-Japan)」
東京大学医科学研究所 システムウイルス学分野の佐藤佳教授が主宰する研究チーム。日本国内の複数の若手研究者・研究室が参画し、研究の加速化のために共同で研究を推進している。現在では、イギリスを中心とした諸外国の研究チーム・コンソーシアムとの国際連携も進めている。
注2)SARS-CoV-2関連コロナウイルス
SARS-CoV-2と系統的に近縁なウイルスのこと。中国およびカンボジア、ラオスといった東南アジアの国々の野生動物から多く発見されている。これらの国々から見つかるウイルスと比べるとSARS-CoV-2と遠縁ではあるが、日本のコウモリからもSARS-CoV-2関連コロナウイルスが検出、分離されている。
注3)異種間伝播(スピルオーバー)
病原体が元来感染している自然宿主から、別の動物種に感染すること。
注4)スパイク(S)タンパク質
コロナウイルスが細胞に感染する際に、細胞に侵入するために必要な構造タンパク質。宿主受容体との結合や細胞融合に関わる重要なタンパク質である。
注5)人工多能性幹細胞(iPSC、iPS細胞)
多様な種類の細胞に分化する能力(多能性)と、無限の増殖性を兼ね備えた細胞。ヒトiPS細胞は、血液や皮膚の細胞などに特定の因子を導入することにより樹立される。
注6)合胞体形成活性
合胞体とは、コロナウイルスに感染した細胞が、スパイクタンパク質を細胞表面に発現し、周囲の細胞と融合することによって形成される大きな細胞塊のこと。合胞体形成活性とは、コロナウイルスのスパイクタンパク質を介して、合胞体を形成する能力のこと。